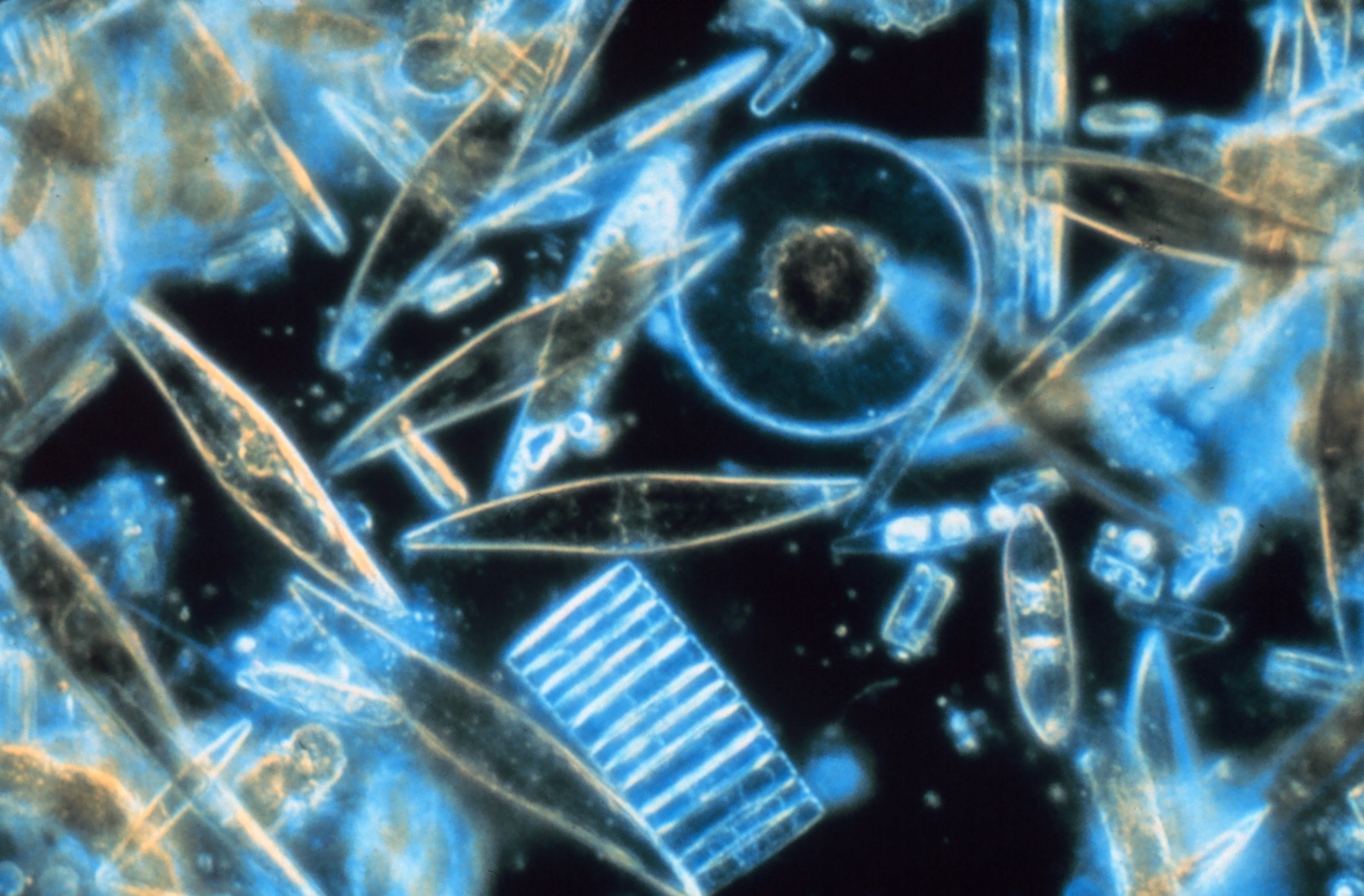
A pesar de los avances técnicos, la mayor parte de las bacterias aún permanecen en el anonimato
Bacterias... Una palabra que nos resulta tan familiar ahora pero que fue un tremendo shock cuando, en el siglo XVII, Anton Van Leeuwenhoek observó unos bichitos invisibles al ojo humano que se encontraban en todas partes, desde los lagos a las afueras de Delf, su ciudad natal, hasta sus propias heces, y que únicamente podía observar con las lentes que cuidadosamente tallaba. Y más impactante debió de ser cuando Pasteur afirmó en el siglo XIX que esos minúsculos seres eran los causantes de la mayoría de las enfermedades. Pero no hay que temer a las bacterias. Las bacterias no son tan diferentes al resto de seres vivos, incluidos nosotros: las hay buenas, las hay malas y las hay simplemente inofensivas. Y sí, la mayoría son inofensivas. Al igual que no tememos que nuestro vecino del tercero sea un asesino y nos apuñale mientras compartimos ascensor, o que nuestras compañeras de piso las hormigas nos devoren sin piedad durante la noche, tampoco debemos temer que haya bacterias en nuestras sábanas. Las hay muy malas, sí, pero no es lo común.
Las bacterias, como ocurre con el resto de seres vivos, se dividen en diferentes especies. Si en el caso de los animales hablamos de perros (Canis lupus familiaris), linces ibéricos (Lynx pardinus) o tigres (Panthera tigris), en las bacterias hablamos de Pelagibacter ubique, Prochlorococcus marinus o Escherichia coli. Y al igual que ocurre con los perros, las bacterias también tienen distintas razas, a las que llamamos cepas.
Si pensamos en las diferentes razas de perros, vemos que todas tienen características comunes que las definen como perros pero, a su vez, cada una tiene una característica diferente que le hace ser mejor en algo: aquellas razas con buen olfato son mejores rastreadores; las hay con buen oído y agresividad con extraños, perfectas como perro guardián; otras obedientes y con sentimiento de pertenencia al rebaño, ideales para pastorear. Con las bacterias pasa lo mismo: sí, hay especies, y sí, hay razas (cepas), cada una con una característica que la hace especial.
Pero, ¿cómo podemos diferenciar a las bacterias? ¿Y cómo podemos saber qué características diferencian a las distintas cepas? Tradicionalmente, al igual que con los perros, las especies de bacterias se clasificaban en función de su forma, gracias al microscopio y, las distintas cepas, en función de si producían o no enfermedad. Sin embargo, esta forma de estudio es muy limitada porque solo permite estudiar las bacterias que se pueden cultivar en el laboratorio, que son muy pocas.
Otra forma de distinguir especies es a través de su ADN: una molécula presente en todas las células que contiene la información necesaria para realizar sus funciones vitales. Cada función se lleva a cabo por un fragmento de ADN llamado gen, y el conjunto de todos los genes de un organismo se llama genoma. Como cada organismo realiza diferentes funciones, su ADN también será diferente. De este modo, si leemos el genoma completo de un organismo y lo comparamos con el genoma de otro organismo de su misma especie, podremos saber si pertenecen a la misma raza o no viendo qué funciones son las que los diferencian.
Pero, ¿no se tarda demasiado tiempo en leer un genoma? El Proyecto Genoma Humano comenzó en el año 2000 y se completó en el año 2003. Tres años para un único genoma. Esto ocurrió porque se hacía de forma lineal, desde el principio hasta el final. Ahora cogemos el ADN, lo hacemos trocitos y leemos cada trozo al mismo tiempo, en paralelo. Así, reducimos el tiempo de años a unas horas. Sin embargo, con las bacterias tenemos un problema añadido: a diferencia del resto de seres vivos, al tratarse de organismos microscópicos es muy difícil separar células individuales. ¿Qué hacemos, entonces?
La solución escogida no es perfecta, pero sí efectiva: estudiar el genoma o conjunto de genes de poblaciones de bacterias en lugar de un individuo en particular. El procedimiento es exactamente el mismo que con organismos por separado: cogemos una muestra, por ejemplo de agua de mar. Rompemos las células que hay en esa muestra, extraemos su ADN, lo hacemos trozos y los leemos en paralelo. El problema ahora es saber a qué bacteria pertenece cada trozo. El procedimiento es el siguiente:
1) Intentamos conseguir trozos de ADN más grandes. Para ello, y teniendo en cuenta que los trozos de ADN solapan entre ellos, unimos las partes que solapan.
2) Una vez que tenemos trozos más grandes, buscamos genes en ellos. Pero, ¿cómo?
Como se explicó anteriormente, los genes son fragmentos de ADN que llevan a cabo una función concreta (por ejemplo, un gen se encarga de que las semillas de la planta del guisante sean amarillas o verdes). El ADN está formado por cuatro letras: A, G, C, T. Los genes comienzan y terminan con unas “palabras” determinadas. Una de esas palabras de inicio de un gen es ATG, y una de las palabras con las que termina un gen es TAG. Así que...
Si en mi fragmento de ADN encuentro la palabra ATG, lo que estará a la derecha de esa palabra será un gen. Y si encuentro TAG, lo que estará a la izquierda de esa palabra será un gen.
ACGTGGCATGCCCTGCGATCGATCATAGTT
Vale, ya tengo el gen. ¿Cómo sé qué gen es? Utilizando una especie de biblioteca donde se guardan todos los genes de todos los organismos que conocemos, y comparando mi gen con ellos. Si se parece lo suficiente a un gen de la biblioteca, sabremos que es el mismo gen (que realiza la misma función) y, además, a qué especie pertenece.
¡Y ya está! Ya tengo los genes de mi población analizados. Pero ahora viene lo divertido: ¿qué pregunta me quiero hacer? ¿qué quiero descubrir? Como dije al principio, cepas o “razas” de bacterias. Más concretamente, quiero saber si una cepa es más abundante en un ambiente que en otro y qué función hace que sea más abundante. Volviendo al ejemplo de los perros, quiero saber si hay más huskies en Siberia que en México y, así, saber que el espeso pelo de los huskies viene muy bien para protegerse del frío. Pero lo voy a hacer de una manera peculiar.
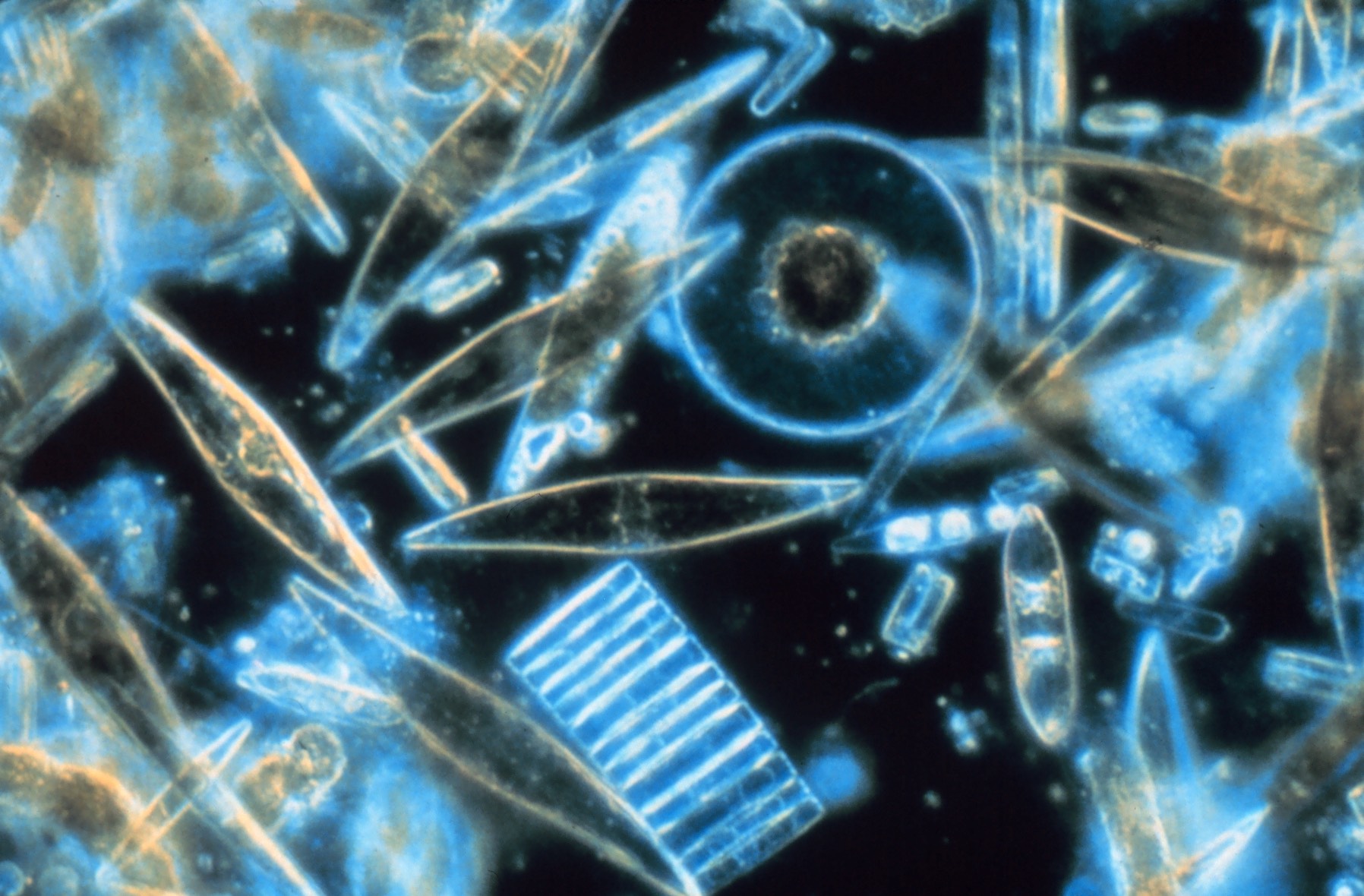 Diatomeas vistas a través de un microscopio, 1983. Autor: Prof. Gordon T. Taylor, Stony Brook University (Public Domain)
Diatomeas vistas a través de un microscopio, 1983. Autor: Prof. Gordon T. Taylor, Stony Brook University (Public Domain)
Si queremos diferenciar razas de perros es sencillo: basta ver a un chihuahua y un husky para saber que son razas diferentes. El problema es que no todas las bacterias se pueden ver porque no todas son cultivables en el laboratorio. Otra forma de hacerlo sería comparar su ADN. Pero no tenemos el ADN completo de una bacteria, sino muchos genes de distintos individuos que pertenecen a la misma especie. Es decir, siguiendo nuestro ejemplo, hay dos poblaciones de perros: los perros de Siberia y los perros de México. Saco el ADN de cada población, lo hago trocitos, busco los genes y meto los genes de cada población en una bolsa. Ahora tengo una bolsa con todos los genes de los perros de Siberia y otra bolsa con todos los genes de los perros de México. Pero, si imaginamos que los perros son invisibles a mis ojos, no sé qué razas hay en cada población ni cuántos perros hay, solo tengo sus genes en dos bolsas. ¿Qué hago?
Muy sencillo: yo sé que todos los perros tienen una sola cola. Y eso es para todos los perros igual. Si cuento el número de colas, sabré el número de perros que hay en cada bolsa. Resulta que en el grupo de Siberia tengo 5 colas de perro y, por lo tanto, 5 perros, y en el de México tengo 8 colas de perro y, por lo tanto, 8 perros.
Ahora miro los genes. Me fijo en los genes que forman el pelo. En el grupo de Siberia tengo 10 genes y en el grupo de México tengo 4. Con una sencilla división:
Siberia:10 genes / 5 perros = 2 genes relacionados con el pelo por perro.
México: 4 genes / 8 perros = 0.5 genes relacionados con el pelo por perro.
¿0.5 genes por perro? Como los genes no se pueden partir, es lógico pensar que la mitad de los perros tienen un gen de pelo y la otra mitad no tienen el gen y están calvos. O también podría pasar que dos perros tuvieran dos genes de pelo y el resto estuvieran calvos.
Es decir, no sabemos cuántos huskies hay en Siberia ni cuántos huskies hay en México. O si son huskies siquiera. Lo que está claro es que en Siberia tener mucho pelo es una ventaja y en México claramente no. Y lo mismo podemos hacer para otros genes, como el gen que determina su tamaño. Cambiemos animales por bacterias y perros por Pelagibacter ubique (la especie de bacteria que me interesa) y tendréis el estudio que estoy haciendo ahora mismo.
Es una pena que no sepamos si son huskies, mastines o labradores. Ni cuántos hay de cada raza. Es lo que ocurre cuando no queda más remedio que recurrir a métodos indirectos. ¿La parte buena? Las dificultades nos hacen desarrollar la creatividad y las nuevas ideas que van apareciendo nunca dejan de sorprendernos. Además, ¿no sería la vida demasiado aburrida si todo fuera tan fácil?